检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
/ 6165金沙总站官方入口 / 学术科研
2019年4月17日,6165cc金沙总站检测中心、蛋白质与植物基因研究国家重点实验室成员陆剑研究员受邀在分子生物学综述类知名期刊《生物化学趋势》(Trends in Biochemical Sciences)发表题为“真核生物上游开放阅读框的功能与演化”(Function and Evolution of Upstream ORFs in Eukaryotes)的综述文章,总结了uORF的最新研究进展以及这一领域当前尚未解决的问题。
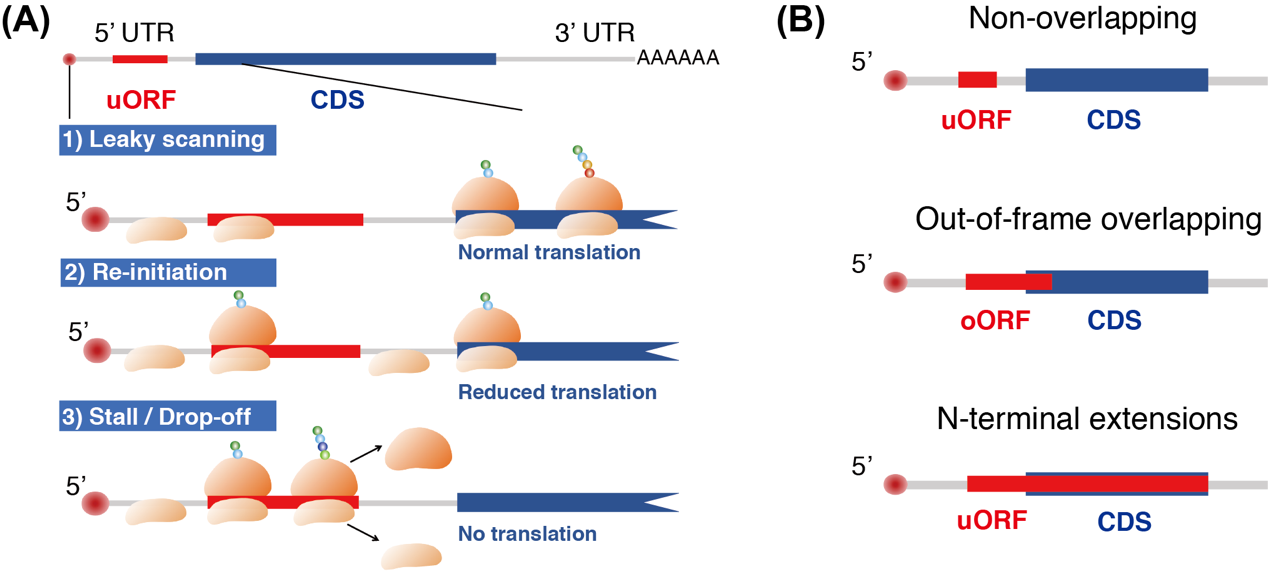
图1. uORF介导的翻译调控。(A)核糖体翻译含有uORF的mRNA时可能发生的三种情况(渗漏扫描、重新起始、停滞/解聚),uORF对CDS翻译起始总体影响表现为抑制作用。(B)uORF的分类。
mRNA翻译调控在真核生物各种生命活动中发挥重要作用。mRNA翻译分为翻译起始、延伸、终止和核糖体的循环利用四个步骤,其中翻译起始是决定mRNA翻译速率的关键步骤。uORF是起始密码子位于CDS上游的开放阅读框,可以通过竞争性结合核糖体来抑制CDS的翻译起始(图1)。uORF在几乎所有真核生物中都广泛存在,转录组中大约一半mRNA的5'非编码区中都含有uORF。然而在过去几十年里,人们对uORF的认识主要基于GCN4和ATF4等少数几个基因,基因组中绝大多数uORF的功能和演化规律还缺乏系统的研究。近年来,随着RNA测序和核糖体图谱(ribosome profiling)技术的发展,越来越多的研究从基因组水平对uORF的功能和演化规律进行系统的解析,极大地推动了这一领域的发展。
陆剑实验室先前在PLoS Biology发表了题为“Genome-wide maps of ribosomal occupancy provide insights into adaptive evolution and regulatory roles of uORFs duringDrosophiladevelopment”的研究论文(Zhanget al.2018),首次揭示了黑腹果蝇基因组中新固定的uORF(上游开放阅读框)受到正向选择,并发现在果蝇发育过程中 mRNA 通过改变 uORF 数目或翻译效率来调控下游蛋白质翻译这一新机制。 在此基础上,陆剑实验室应邀撰写综述总结了近年来关于uORF的基因组水平研究,指出真核生物基因组中大多数uORF都能够被核糖体翻译,uORF对CDS的调控作用受到uORF本身以及周围序列特征的影响;基因组中uORF的分布是正向选择和纯化选择共同作用的结果;uORF介导的翻译调控在生物体的生长发育、胁迫应答和肿瘤发生等过程中发挥重要作用(图2)。同时该综述还分析了这一领域尚未解决的问题,为未来的研究提供了可能的方向。
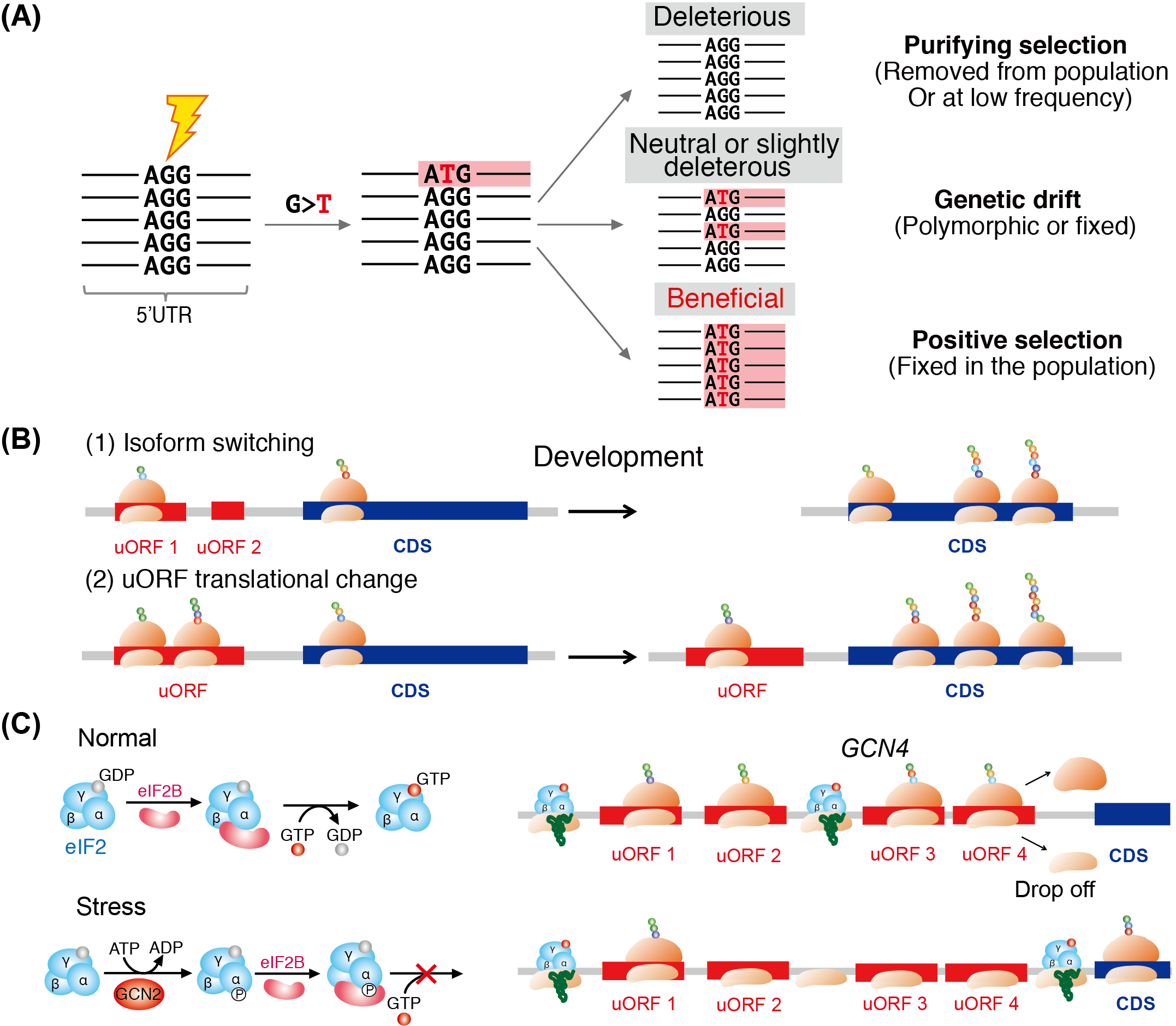
图2. uORF的演化规律和生物学功能。(A)新产生的uORF可能受到不同的演化驱动:有害的uORF会被自然选择很快从群体中清除,中性的或者轻微有害的uORF会在群体中随机漂变;有利的uORF会在自然选择作用下在群体中很快固定,并在基因组中维持下来。(B)在发育过程中,mRNA可以通过改变5'非编码区uORF的组成或者uORF的翻译效率来调控CDS翻译。(C)uORF在正常情况下抑制GCN4CDS的翻译。在胁迫条件下,eIF2的a亚基被磷酸化,导致翻译完uORF1的核糖体在重新起始过程中遗漏其他的uORF而在CDS处起始翻译,促进GCN4的表达。
陆剑实验室致力于利用比较基因组学和核糖体图谱等功能基因组学方法,系统解析参与翻译调控的顺式和反式元件的进化规律和生物学功能。围绕“mRNA翻译调控元件的进化驱动及其在适应性进化中的作用”这一主题,陆剑研究员课题组近年来在PLoS Biology、Nucleic Acid Research、Nature Communications、Molecular Biology and Evolution等国际主流期刊发表了一系列研究成果(全文链接见https://lujianlab.org/publications.html)。本次特邀综述的发表,体现出陆剑实验室在mRNA翻译调控领域的研究得到了国际同行的关注和认可。
陆剑研究员为该综述文章的通讯作者,其课题组的博士生张宏和博士后王奕蓉为共同第一作者。此项工作得到了国家自然科学基金委重大研究计划“微进化过程的多基因作用机制”项目和科技部等的资助。
文章链接:
https://www.cell.com/trends/biochemical-sciences/fulltext/S0968-0004(19)30055-6
https://journals.plos.org/plosbiology/article/authors?id=10.1371/journal.pbio.2003903