检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
/ 6165金沙总站官方入口 / 学术科研
新型冠状病毒肺炎(COVID-19)的爆发已经在全球范围内引发了严重的公共卫生危机。SARS-CoV-2是导致COVID-19的主要病原体,它是继SARS-CoV和MERS-CoV之后第三种对人类具有高致病性的冠状病毒[1]。研究表明,ACE2(血管紧张素转化酶2)是介导SARS-CoV-2入胞的高亲和力受体,SARS-CoV-2通过其表面刺突蛋白(Spike,S)的受体结合域(RBD)与ACE2互作而感染细胞。然而,ACE2在人体器官中的表达分布与SARS-CoV-2的器官嗜性并不完全相关。比如,在整个呼吸道、大脑以及多种免疫细胞中,ACE2的表达较低或几乎不表达,但是这些细胞仍然可以被SARS-CoV-2感染。因此,可能存在ACE2以外的受体来介导病毒进入宿主细胞。
目前,已有多项研究发现与SARS-CoV-2感染相关的关键宿主因子,比如基于CRISPR基因敲除系统的高通量功能性筛选。然而,这些筛选均没有发现ACE2以外的新受体,这可能是因为此类基于功能缺失的筛选是在ACE2的表达和功能占主导地位的细胞类型中进行的[2-5]。
2021年8月20日,北京大学魏文胜课题组、中国医学科学院/北京协和医学院王健伟课题组与北京大学肖俊宇课题组在Science China Life Sciences杂志联合发表了题为Genome-wide CRISPR activation screen identifies candidate receptors for SARS-CoV-2 entry的研究论文,该研究通过基于CRISPR激活(CRISPR activation, CRISPRa)系统的全基因组水平的功能获得性筛选,发现了多个介导SARS-CoV-2入侵细胞的潜在新受体。
为了系统性研究SARS-CoV-2入胞的关键因子,该研究使用SARS-CoV-2假病毒在HEK293T细胞中进行了全基因组CRISPRa筛选。该筛选基于实验室之前建立的内置分子条形码(iBAR)方法[6]在sgRNA中添加外置条形码(eBAR),建立sgRNAeBAR文库,从而可以使用高感染复数及较少的细胞量进行高质量文库构建。筛选结果表明,除了发现已知的SARS-CoV-2受体ACE2、主要宿主细胞蛋白酶TMPRSS2以及已报道的ACE2依赖性共受体NRP1外,还鉴定出多种新型宿主因子可能参与SARS-CoV-2入胞机制中(图1)。
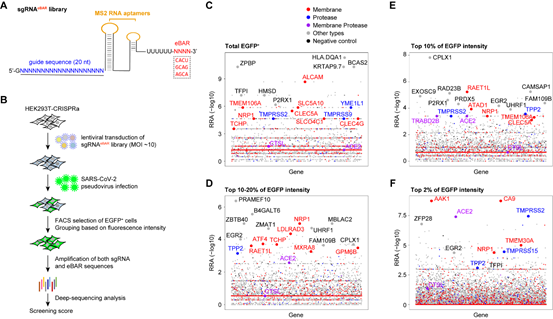
图1 通过全基因组CRISPRa功能性筛选发现影响SARS-CoV-2入胞的关键宿主因子
结合SARS-CoV-2假病毒和真病毒实验,该研究发现膜蛋白LDLRAD3、TMEM30A和CLEC4G能够以不依赖于ACE2的方式有效介导病毒入侵细胞。此外,研究证实这些膜蛋白都能够与SARS-CoV-2 S蛋白结合。与ACE2结合S蛋白的RBD不同,这些蛋白能够特异性结合S蛋白的N末端结构域(NTD)(图2)。LDLRAD3在神经元中高度表达,最近作为委内瑞拉马脑炎病毒(VEEV)的关键受体受到关注。实验表明,敲低LDLRAD3或在细胞上清中加入其可溶性蛋白可显著降低SARS-CoV-2对神经元细胞的感染。CLEC4G在肝脏、淋巴结和单核细胞中高度表达,已知其作为细胞粘附因子促进SARS-CoV感染宿主;通过在肝脏细胞中进行敲低实验证实了CLEC4G在SARS-CoV-2入侵细胞中的重要作用。跨膜蛋白TMEM30A在最近的SARS-CoV-2全基因组敲除筛选(Huh7.5细胞)中也被鉴定出来[4],研究进一步证实了其对于SARS-CoV-2入胞的重要性以及其与S蛋白的直接结合。这些具有组织特异性或广谱表达的新受体的发现,有助于进一步揭示SARS-CoV-2的多器官嗜性,并为探究COVID-19的新治疗靶点提供线索。
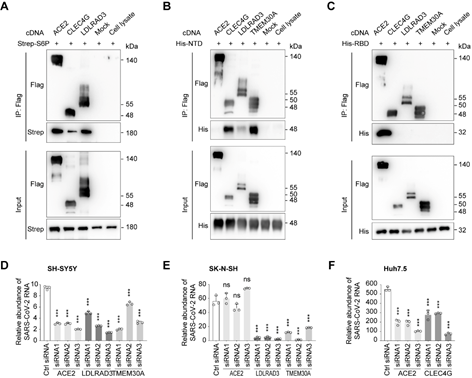
图2 SARS-CoV-2感染实验证实LDLRAD3、CLEC4G和TMEM30A为介导SARS-CoV-2入胞的潜在受体
北京大学魏文胜课题组博士后朱诗优博士、刘莹博士、副研究员周卓博士和肖俊宇课题组博士生张志莹为该论文的共同第一作者。该研究项目得到了国家重点研发计划、国家自然科学基金、传染病防治国家科技重大专项、北京市科委生命科学前沿创新培育项目、北京未来基因诊断高精尖创新中心、北大-清华生命科学联合中心以及中国博士后科学基金的支持。
原文链接:http://engine.scichina.com/doi/10.1007/s11427-021-1990-5
参考文献:
1. Andersen, K.G., Rambaut, A., Lipkin, W.I., Holmes, E.C., and Garry, R.F. (2020). The proximal origin of SARS-CoV-2. Nat Med 26, 450-452.
2. Wei, J., Alfajaro, M.M., DeWeirdt, P.C., Hanna, R.E., Lu-Culligan, W.J., Cai, W.L., Strine, M.S., Zhang, S.M., Graziano, V.R., Schmitz, C.O., et al. (2021). Genome-wide CRISPR Screens Reveal Host Factors Critical for SARS-CoV-2 Infection. Cell 184, 76-91 e13.
3. Daniloski, Z., Jordan, T.X., Wessels, H.H., Hoagland, D.A., Kasela, S., Legut, M., Maniatis, S., Mimitou, E.P., Lu, L., Geller, E., et al. (2021). Identification of Required Host Factors for SARS-CoV-2 Infection in Human Cells. Cell 184, 92-105 e116.
4. Baggen, J., Persoons, L., Vanstreels, E., Jansen, S., Van Looveren, D., Boeckx, B., Geudens, V., De Man, J., Jochmans, D., Wauters, J., et al. (2021). Genome-wide CRISPR screening identifies TMEM106B as a proviral host factor for SARS-CoV-2. Nat Genet. 53(4), 435-444.
5. Zhu, Y., Feng, F., Hu, G., Wang, Y., Yu, Y., Zhu, Y., Xu, W., Cai, X., Sun, Z., Han, W., et al. (2021). A genome-wide CRISPR screen identifies host factors that regulate SARS-CoV-2 entry. Nature communications 12, 961.
6. Zhu, S., Cao, Z., Liu, Z., He, Y., Wang, Y., Yuan, P., Li, W., Tian, F., Bao, Y., and Wei, W. (2019). Guide RNAs with embedded barcodes boost CRISPR-pooled screens. Genome Biol 20, 20.