检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
/ 6165金沙总站官方入口 / 学术科研
2022年9月22日,上海交通大学医学院李华兵研究员、6165cc金沙总站检测中心伊成器教授、陆军军医大学吴玉章和耶鲁大学医学院Richard Flavell团队合作在Nature Immunology上发表了题为 tRNA-m1A modification promotes T cell expansion via efficient MYC protein synthesis 的研究论文,该研究揭示了m1A修饰催化酶TRMT61A通过催化新合成tRNA第58位腺嘌呤的1-甲基腺嘌呤(m1A)修饰,加速CD4+ T细胞激活后多种关键蛋白的翻译,保证CD4+ T细胞的迅速免疫应答。

T细胞在免疫反应中发挥核心作用。当受抗原刺激时,处于静息状态的T细胞快速激活,大量增殖分化。免疫激活是免疫反应中非常重要的一步,之前的研究集中于探究转录调控在T细胞激活过程中的作用[1]。鉴于T细胞激活在短时间内快速发生,转录调控不能及时满足T细胞大量合成蛋白的需求,因此翻译效率的调控可能也发挥重要作用。
tRNA在翻译过程中发挥着重要作用。除了tRNA丰度之外,tRNA上的化学修饰可以通过稳定tRNA结构,帮助tRNA进行codon识别等途径影响翻译过程。其中,m1A是tRNA上丰度最高最保守的一种修饰,由TRMT6/TRMT61A复合物进行修饰,tRNA-m1A修饰可以增强翻译起始和延伸 [2]。伊成器课题组在2017年建立了m1A修饰位点的单碱基分辨率鉴定技术[3]。
该研究首先观察到T细胞激活过程中蛋白质翻译相关通路上调,不同的tRNA也表现出不同时间点上调的动态表达模式,tRNA-m1A58修饰酶TRMT6/TRMT61A在激活过程中也上调。作者利用Trmt61a条件性敲除小鼠,证明在体内、体外条件下,Trmt61a缺失时T细胞激活免疫功能受损,T细胞增殖能力显著降低。同时,发现Trmt61a缺失导致T细胞中大部分tRNA的m1A58修饰显著减少,导致 T细胞激活后多种关键蛋白的翻译受阻,特别是转录因子MYC。MYC蛋白翻译效率的降低使T细胞激活后的快速克隆扩增受阻。
作者随后探究了Trmt61a调控翻译的特异性。通过对RiboTag-seq, tRNA-seq,tRNA-m1A-seq的多组学联合分析,研究人员发现翻译效率下调的基因(如Myc)有tRNA codon使用偏好性,更偏好使用在激活早期高表达的tRNA,而这部分tRNA又是Trmt61a缺失高度敏感的tRNA。
该研究第一次将tRNA修饰与T细胞的功能变化连接起来,并系统性地探究了原代T细胞扩增过程中控制翻译的具体机制,表明tRNA的使用及其修饰可以对蛋白表达和细胞功能带来显著改变。这项研究表明TRMT61A介导的tRNA-m1A58修饰作为CD4+ T细胞增殖调控的新型“翻译检查点”,将为改善CD4+ T细胞介导的炎症应答及增强肿瘤免疫治疗效果提供新的RNA表观遗传策略。
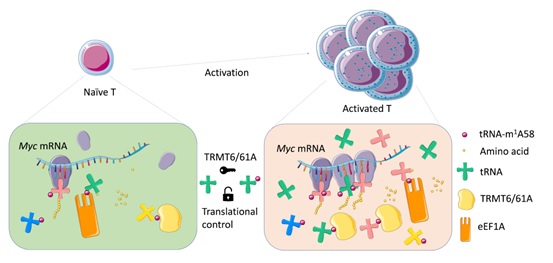
TRMT61A介导的tRNA-m1A58修饰是CD4+ T细胞激活增殖调控的“翻译检查点”
上海交通大学医学院刘永波博士、周静副研究员和硕士研究生施金彤,6165cc金沙总站检测中心已出站博士后李笑雨(现浙江大学医学院研究员),博士研究生张晓婷为本论文的共同第一作者,上海交通大学医学院李华兵研究员、6165cc金沙总站检测中心伊成器教授、陆军军医大学吴玉章教授及耶鲁大学医学院Richard A.Flavell教授为该文的共同通讯作者。
原文链接:https://www.nature.com/articles/s41590-022-01301-3
参考文献:
1.Kaech, S. M. & Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nat Rev Immunol 12, 749–761 (2012).
2.Liu, F. et al. ALKBH1-Mediated tRNA Demethylation Regulates Translation. Cell 167, 816-828.e16 (2016).
3. Li, X. et al. Base-Resolution Mapping Reveals Distinct m1A Methylome in Nuclear- and Mitochondrial-Encoded Transcripts. Mol. Cell 68, 993-1005.e16 (2017).